谱聚类再探
本文最后更新于 2024年4月9日 下午

首先查阅相关的资料,比较谱聚类算法与传统的聚类算法K-mean的区别。
K-mean
K-mean实现:
- 确定K值,随机选取K个点
- 以这K个点为中心质点,对原始数据集中中的每个点分别计算离哪个中心点更近
- 计算结束就将原始数据集分为K个类,对K个类重新计算中心质点(平均值法)
- 若全部K类新计算出的中心质点与该类上一个中心质点的距离小于一个阈值,则认为聚类操作已经收敛了,聚类结束
- 否则重复步骤2-4
K-mean的优缺点主要如下:
- 优点:
- 原理简单,易于理解
- 聚类效果不错
- 缺点:
- 需要提前确定K值(划分种类数)
- 个别噪点对于总体效果影响较大
Spectral Clustering
需要先了解:
- 邻接矩阵W:衡量两个点之间关系(比如位置上接近,表示两个点关联紧密)的一个矩阵,常有很多的衡量方法。
- 度矩阵D:是一个对角矩阵,对角线上每一行元素的值为邻接矩阵W中每行元素值的和。
- 拉普拉斯矩阵L:该矩阵具有很多的性质,谱聚类算法与这些性质密切相关
- 拉普拉斯矩阵是一个半正定矩阵
- 对于任何向量L下列式子成立

主要思想:
如果有A和B两个子图,两个子图间的联系是不是可以用下面这种方式来表示呢?
我们只要使上面的式子最小化,是不是就可以保证两个子图联系最小,就可以认为这是两个不相关的部分。
但是如果单纯依靠这种方式,会导致倾向于将孤立的点切开,因为单独一个点到其他部分的权值肯定是最小的。怎么避免这种问题出现呢?有两种办法:
-
一是将最小化目标改成如下:
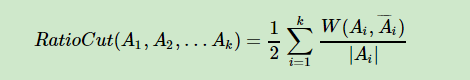
-
而是将最小化目标改为:
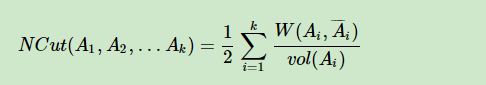
这也就引出了谱聚类的两种常用方法。
但是怎么最小化这个值呢?
我们以第一个RdtioCut为例。大神们发现,可以引入一个向量 $h_j (1<=j<=k)$ ,这个向量表示的是什么呢?
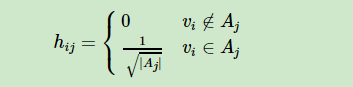
向量 $h_j$中的每个值 $h_{ij} (1<=i<=n)$ 有两种取值,表示坐标点 $i$ 是不是属于子图 $A_j$ ,$|A_j|$ 表示子图中节点数量
对这个向量进行如下的运算:
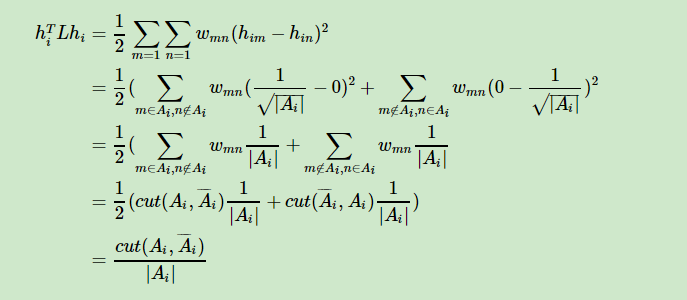
我们会惊奇的发现这不就是我们最小化的目标吗?只不过现在图中的点只分为了两部分(属于$A_i$ 和不属于)
如果我们想将图分为更多的子图,那么我们的目标就成为了:划分K个子图,每个子图和子图以外部分的连接权重和最小。也即使如下公式最小:
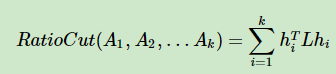
我们还知道向量 $h_i$和$h_j$是表示点划分的两种情况,$h_i$表示属于$A_i$的点,$h_j$表示属于$A_j$的点。那么这两个向量就应该是正交的。并且 $ h^T h = 1 $。这时我们引入瑞丽商的性质:
- 当 $h_i^T * h_i=1 $
- $h_i^T L h_i$等于L的特征值
也就意味着我们最小化的目标变为了求:$H^T L H (H={h_1,h_2…h_k})$ 这个式子最小的k个特征值,他们对应的就是划分最准确的情况。但还有一个问题,我们得到了最小的K个特征值和他们对应的特征向量,怎能才能利用他们聚类呢?其实,我们可以把所得特征值向量对每个样本的表示看作是一种类别编码,我们通过传统聚类的方法能够得到最终结果。
另外
对于Ncut稍不同于上面的RatioCut。因为Ncut中,虽然也引入了一个向量$h_i$,但是向量定义为如下,$vol(A_j)$ 表示的是子图中节点度的和。
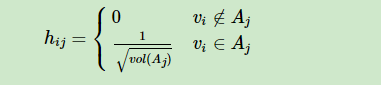
然后步骤都类似于RatioCut只不过在利用瑞丽商的时候出现了问题,因为 $ h^T h = 1 $并不成立了。所以需要采取一些办法,根据
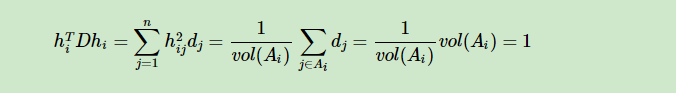
$m_i^T m_i=1 $
$m_i=D^{1/2}*h_i$
我们需要把现在的 $h_i$ 变成 $m_i$
也即 $h_i^T L h_i = m_i^T * D^{-1/2} * L * D^{-1/2} * m_i =h_i^T * D^{1/2} * D^{-1/2} * L * D^{-1/2} * D^{1/2} * h_i$
此时要求特征值的矩阵变为: $D^{-1/2} * L * D^{-1/2}$
谱聚类实现步骤:
- 定义邻接矩阵W
- ε 近邻法
- 只取距离小于ε的边,否则为0
- K 近邻法
- 两点互为K近邻,否则为0
- 其一为K近邻,否则为0
- 全连接法
- 常采用高斯核函数的方法,每两个点之间的权值都不为0
- ε 近邻法
- 度矩阵 D
- 可根据邻接矩阵W得
- 拉普拉斯矩阵矩阵 L
- $L= D - W$
- 正规化
- $D^{-1/2} L D^{-1/2}$
- 计算特征向量
- 计算K个最小特征值对应的特征向量$h_i$,特征向量组成矩阵$H^{n*k}$
- H标准化
- 消除奇异值带来的影响
以上是谱聚类第一大部分,降维
- H使用K-mean进行最后的聚类
- 得出结果
这是谱聚类的第二大部分,聚类
NCut 推导细节
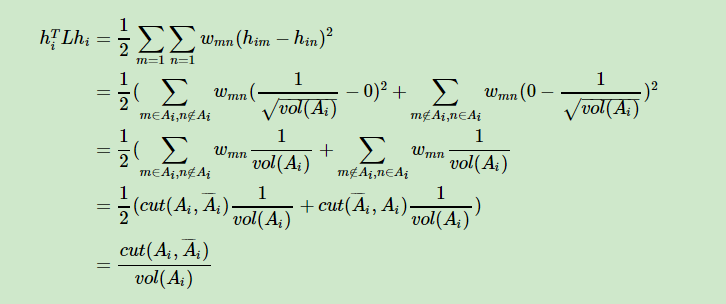
RadioCut推导细节
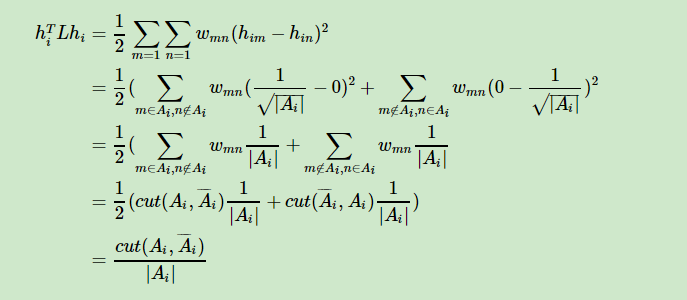
谱聚类优势
- 效果好
- 由于采用了降维的思想,处理高维数据时有优势
实现代码
1 | |
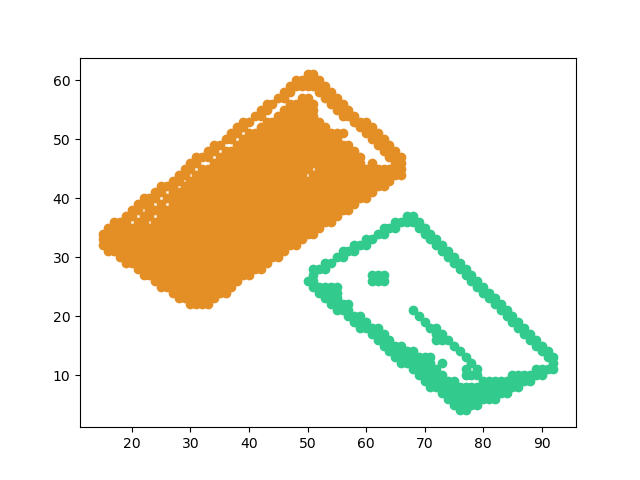
效果
原图:

聚类后:
![[公式]](/img/loading.gif)